
Google語音識別技術詳解與實踐應用
小杜的生信筆記,自2021年11月開始做的知識分享,主要內容是 R語言繪圖教程 、 轉錄組上游分析 、 轉錄組下游分析 等內容。凡事在社群同學,可免費獲得自2021年11月份至今全部教程,教程配備事例數據和相關代碼,我們會持續更新中。
聚類分析用于將表達模式相同或相近的基因聚集成類,進而識別未知基因的功能或已知基因的未知功能,這些同類基因可能具有相似的功能,共同參與同一代謝過程或存在于同一細胞通路中。K-means稱為K-均值聚類;k-means聚類的基本思想是根據預先設定的分類數目,在樣本空間隨機選擇相應數目的點做為起始聚類中心點;然后將空間中到每個起始中心點距離最近的點作為一個集合,完成第一次聚類;獲得第一次聚類集合所有點的平均值做為新的中心點,進行第二次聚類;直到得到的聚類中心點不再變化或達到嘗試的上限,則完成了聚類過程。
在多組學分析中,趨勢圖是常見的圖形之一,而聚類分析則是構建這類圖形的基礎。通過聚類,我們可以識別相似表達模式的基因,進而進行更深入的生物學解讀。
ClusterGVisClusterGVis 包可以使用 k-means 或 mfuzz 進行聚類分析。具體操作,自己動手做一下即可。
一般,我們輸入的都是 寬數據矩陣,如下所示:
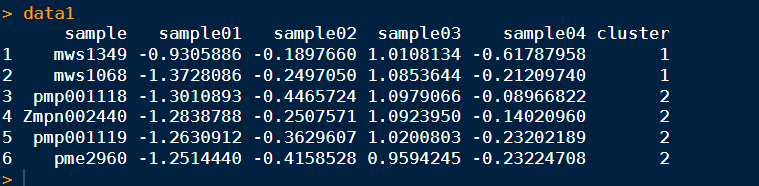
cluster 是我們已經做好分類的列。
data2 <- pivot_longer(data1, cols = -c(sample, cluster), names_to = "group", values_to = "value")data2 <- data2[, c("sample", "value", "group", "cluster")]
data2df2 %
group_by(cluster, group) %>%
summarise(value = mean(value))
df2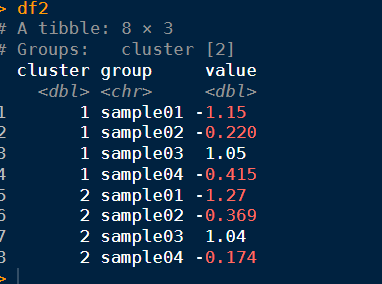
ggplot(data2, aes(x = group, y = value)+)
gm_line(aes(group = sample), color = "grey90", size = 0.5)+
##'@X軸因子固定,結合自己的數據進行修改
scale_x_discrete(limits = c("sample1", "sample2", "sample3", "sample4", "sample5")) +
geom_line(data = df2, aes(x = group, y = value, group = 1), color = "red", size = 1)+
facet_wrap(~ factor(cluster), nrow = 2)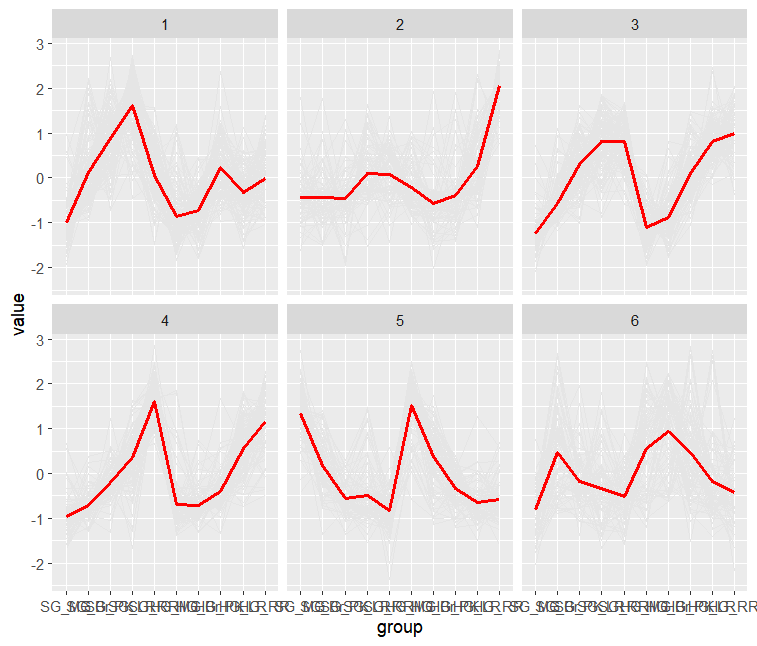
ggplot(data2, aes(x = group, y = value)+)
gm_line(aes(group = sample), color = "grey90")+
##'@X軸因子固定,結合自己的數據進行修改
scale_x_discrete(limits = c("sample1", "sample2", "sample3", "sample4", "sample5"))+
stat_summary(aes(group = 1), fun.y = "mean", geom = "line", size = 1, color = "red")+
theme_classic(base_size = 14)+
theme(axis.ticks.length = unit(0.1,'cm'),
axis.text.x = element_text(angle = 45,
hjust = 1,color = 'black'),
strip.background = element_blank())+
facet_wrap(~factor(cluster), nrow = 2)+
ylab('Normalized expression') + xlab(NULL)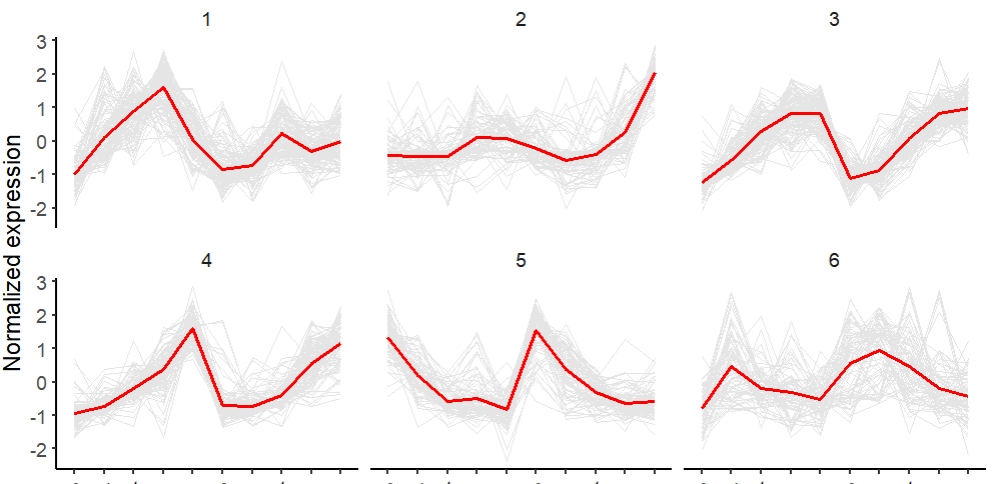
圖形其余美化,結合自己的需求進行調整即可。
聚類分析在基因表達數據中可以用于鑒定共表達基因,這對于理解基因的功能和調控網絡至關重要。
通過聚類分析,我們可以將表達模式相似的基因聚為一類,進而分析這些基因的生物學功能。
在蛋白質組學中,聚類分析同樣重要,可以幫助我們識別參與相同生物學過程的蛋白質。
通過分析蛋白質表達模式的聚類,我們可以識別在特定生物學過程中起關鍵作用的蛋白質。
答:K-means聚類是一種將數據點分成K個簇的算法,使得簇內的點盡可能相似,簇間的點盡可能不同。
答:選擇合適的K值可以通過多種方法,如肘部法則、輪廓系數等,這些方法可以幫助我們評估不同K值下的聚類效果。
答:K-means聚類的優點包括簡單、高效,適用于大規模數據集;缺點包括對初始質心敏感,可能需要多次運行以獲得穩定結果,且對噪聲和離群點敏感。
答:處理離群點可以采用多種策略,如在聚類前預處理數據以識別和移除離群點,或者使用對離群點不敏感的聚類算法,如K-medoids。
答:聚類分析在生物信息學中有廣泛的應用,包括基因表達數據的分析、蛋白質組學數據的分析、代謝組學數據的分析等,可以幫助我們識別共表達的基因、關鍵蛋白質和重要代謝物。